Różnicowanie fenotypowe i genotypowe drożdży z rodzajuCandidaizolowanych z kału zwierząt mięsożernych (psów i kotów)
Różnicowanie fenotypowe i genotypowe drożdży z rodzaju Candida izolowanych z kału zwierząt mięsożernych (psów i kotów).
Zakład Mykologii1, Zakład Parazytologii i Inwazjologii2, Katedra Nauk Przedklinicznych, Szkoła Główna Gospodarstwa Wiejskiego, ul. Ciszewskiego 8, 02-786 Warszawa.
Altrych Paulina1 Krutkiewicz Alicja1 Klockiewicz Maciej2 Dworecka-Kaszak Bożena1.
Wstęp Grzyby z rodzaju Candida sp. mogą występować u osobników zdrowych jako składnik stałej, endogennej bioty, zasiedlając głównie błony śluzowe organizmu, w tym również przewodu pokarmowego. Wśród komensalnych gatunków znajdują się także potencjalnie chorobotwórcze C.albicans, C.parapsilosis czy C.tropicalis. Obok tradycyjnych metod fenotypowego różnicowania gatunków stosuje się coraz bardziej powszechne metody oparte o różnice w genomie. Najczęściej sekwencjonowane obecnie fragmenty DNA grzybów to Regiony ITS (Internal Transcribed Spacer), charakteryzujące się dużą zmiennością wewnątrzgatunkową i stanowiące podstawę systematyki molekularnej na poziomie gatunku.
Materiał i metody Próbki kału pozyskiwano od psów i kotów. Z każdej próbki wykonywano preparat bezpośredni celem obserwacji obecności komórek drożdżopodobnych oraz wykonywano posiew na podłoże mykologiczne Sabouraud, inkubowano w temp. 30°C przez 72 godziny.
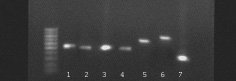
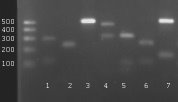
Hodowle oceniano na podstawie makroskopowych cech kolonii oraz mikroskopowej morfologii blastospor. Wykonywano test filamentacji, wynik dodatni uważano za przynależność do gatunku C.albicans. Identyfikację fenotypową przeprowadzono na podstawie testu API CANDIDA (bioMerieux). Przy użyciu zestawu do izolacji DNA z drożdży Genomic Mini AX YEAST wyizolowano DNA ze wszystkich szczepów o potwierdzonej przynależności do z rodzaju Candida. Stosując uniwersalne dla grzybów startery ITS1 – 5’ TCCGTAGGTGAACCTGCGG 3’ ITS4 – 5’ TCCTCCGCTTATTGATATGC 3’ przeprowadzono reakcję amplifikacji. Uzyskano produkt zawierający region ITS1, gen rDNA 5,8S oraz ITS2, który wizualizowano przeprowadzając elektroforezę w 0,8% żelu agarozowym. Porównywano wielkość uzyskanych amplikonów w obrębie jednego gatunku, jak również analizowano różnice w wielkości uzyskanego produktu w zależności od gatunku, z którego pochodziło DNA. Przeprowadzono trawienie uzyskanych produktów reakcji amplifikacji celem rozróżnienia poszczególnych gatunków.
Do analizy restrykcyjnej użyto enzymów DdeI i HpaII dobranych na podstawie analizy sekwencji nukleotydów. Produkty trawienia rozdzielono elektroforetycznie i przeanalizowano różnice we wzorach prążków.
Wyniki Przebadano 723 próbki kału, z czego 348(48%) pochodziło od psów, a 375(52%) od kotów. Z 238(33%) próbek wyizolowano szczepy wstępnie zakwalifikowane do rodzaju Candida sp. Na podstawie cech fenotypowych wykazano obecność innych grzybów drożdżopodobnych – Malassezia sp.5(0,7%), Rhodotorulla sp.47(6,5%) oraz Geotrichum sp.41(5,7%) Na podstawie testów API CANDIDA zidentyfikowano szczepy z rodzaju Candida sp. 128(85,3%): C.albicans 71(47,3%), C.parapsilosis 14(9,3%), C.krusei 12(8%), C.glabrata 5(3,3%), C.tropicalis 3(2%), C.kefyr 3(2%), C.lusitaniae 2(1,3%), C.non-albicans 7(4,6%), C.krusei/parapsilosis/Geotrichum 3(2%), C.lusitaniae/tropicalis/famata 2(1,3%), C.famata/glabrata 2(1,3%), C.famata/guillermondi 1(0,6%), oraz z gatunków Saccharomyces cerevisiae 16(10,6%), Trichosporon spp. 5(3%), Cryptococcus neoformans 1(0,6%). Wyizolowano DNA ze wszystkich 128 szczepów i po przeprowadzeniu reakcji amplifikacji z użyciem odpowiednich starterów analizowano różnice w wielkości uzyskanych produktów. Przeprowadzona analiza restrykcyjna umożliwiła różnicowanie gatunków na podstawie analizy wielkości prążków. Na podstawie wielkości amplikonu będącego produktem reakcji PCR z użyciem uniwersalnych starterów ITS1 i ITS4 możemy jednoznacznie zidentyfikować 3 gatunki: C.glabrata (881pz), C.kefyr(721pz), C.lusitaniae(341pz), wzory prążków należących do poszczególnych izolatów z gatunków C.albicans(536pz), C.krusei(509pz), C.parapsilosis(546pz), C.tropicalis(554pz) wykazują minimalne różnice– co obrazuje ryc.1. Jednakże analiza restrykcyjna z użyciem odpowiednich enzymów pozwala na różnicowanie gatunkowe. Po przeprowadzeniu trawienia z użyciem enzymów DdeI i HpaII uzyskano prążki o wielkościach: C.albicans (297pz,121pz,118pz), C.krusei (260pz,249pz), C.parapsilosis (546pz), C.glabrata (465pz,320pz,51pz,45pz), C.tropicalis (367pz,118pz,69pz), C.lusitaniae (242pz,98 pz), C.kefyr (549pz,172pz) – co obrazuje ryc.2. Wśród zebranych 3 izolatów
zakwalifikowanych do gatunku C.famata ustalonego na podstawie testu API CANDIDA, zaobserwowano różnice w wielkość fragmentu zawierającego region ITS1, gen rDNA 5,8S oraz ITS2, po zsekwencjonowaniu analizowanego fragmentu zakwalifikowano dwa szczepy do gatunku C.lusitaniae, jeden do gatunku C.lipolityca co wskazuje na małą przydatność testu API CANDIDA do różnicowania tego gatunku.
Wnioski:
- Grzyby z rodzaju Candida sp. mogą występować w kale psów i kotów nie wykazujących objawów klinicznych ze strony przewodu pokarmowego.
- Najbardziej rozpowszechnionym gatunkiem jest C.albicans.
- Fenotypowa Klasyfikacja grzybów z rodzaju Candida nie zawsze jest zgodna z identyfikacją genotypową.